MASCOT ver.2.7 における variable modification
この記事では、varable modification に関してMascot がver.2.7 より新設したパラメータについてご紹介します。
パラメータの説明の前に、ver.2.6との違いについても改めて確認いたします。
MASCOT ver.2.6 以前における variable modification の取り扱い
以下のようなルールが適用されていました。
- 「1ペプチドの中で修飾をうけるアミノ酸残基数」に上限値はない
- 修飾の組み合わせパターンについては、組み合わせ総数が16通りを超えるかどうかで適用ルールを変えていました。16通り未満の場合はすべての可能性を考慮しますが、16通りを超える場合はすべての可能性を考慮していませんでした。
総組み合わせ数により検証方法が切り替わる点と、16通りを超える時に考慮できない修飾パターンがあるという事が問題点でした。
MASCOT ver.2.7 から採用された variable modification の取り扱い
MASCOT ver.2.7では、variable modification検索に関する3つのパラメータが導入されました。
| 項目名 | 説明 | 初期設定値 |
|---|---|---|
| MaxPepNumVarMods | 1ペプチドにおいて考慮するVariable modification(種類)数の最大値: | 3 |
| MaxPepNumModifiedSites | 1ペプチドにおいて考慮する、Variable modificationを考慮するアミノ酸残基数の最大値 | 5 |
| MaxPepModArrangements | Variable modification の組み合わせパターンについて考慮する組み合わせ数の最大値 | 64 |
パラメータはmascot.dat 内の記述としてグローバルな(MASCOT Server 全体に及ぶ)設定内容として適用する事ができます。また、各検索ごとにMGF ファイルに組み込ませてローカルな(該当検索でのみ適用される)設定内容として適用することもできます。
そして、総組み合わせ数により評価方法を切り替えることをやめ、定められた範囲内ですべての修飾組み合わせパターンを考慮するようになりました。
これらのパラメータを変更するメリットについては、以下の2通りが考えられます。
- 考慮するパターンを減らし、計算速度を上げるケース (Example 1 へ)
- 考慮するパターンを増やし、複雑な修飾解析に対応させるケース (Example 2 へ)
Example 1: Error Tolerant 検索を速くする
レポジトリサイト PRIDE に登録されている 、生物種がマウスのラベルフリー定量のデータPXD013086を使った解析例をご紹介します(ただし定量解析部分は今回の話と関係ありません)。
データをダウンロードしMASCOT Distiller を使ってデータ処理(ピーク抽出)を行って、2万程度の query (MS/MS データセット)を取得できました。PRIDEで登録されているvariable modification 情報では、OxidationとDeamidation の2つのみがあげられています。このデータについて、MASCOT ver.2.6 と2.7で検索を行いその内容と計算速度について、Error Tolerant検索で比較しました。
ver.2.7 についてはvariable modification に関する設定の値を小さくして検索も試してみました。その時の検索時間についてまとめたデータが table 2 です。
| Mascot Version | ModArrangements | NumModifiedSites | NumVarMods | Search time (min) | Speed improvement (%) |
|---|---|---|---|---|---|
| 2.6 | N/A | N/A | N/A | 95 | — |
| 2.7 | 64 | 5 | 3 | 79 | 17 |
| 2.7 | 32 | 3 | 3 | 61 | 35 |
すべてのケースにおいて最初の検索は2分程度なので、検索時間のほとんどは2段階目の error tolerant 検索段階という事になります。
同定結果という面ではほぼ同様の内容でした:ver.2.6 では 4437 の PSMs * 、Error Tolerant で新たに追加された629のPSMs が、ver.2.7 では 4442 の PSMs, Error Tolerant で新たに追加された635のPSMs が得られました。
*1 PSM: (Peptide Spectrum Matches,ここでは「同定スペクトル数(同一ペプチドへのアサインをまとめずにカウント)」と同義
このケースのように、考慮すべき variable modification 数が少なくても済む場合、variable modificationに関するパラメータの数字を小さくして検索範囲を狭め、検索時間を短縮することができます。
Example 2 : 組み合わせパターンの多い修飾解析
レポジトリサイト PRIDE に登録されている ミドルダウンプロテオミクス用のデータで、Humanのヒストン H4 のデータPXD008296を例としてご紹介します。この解析では、細胞周期における修飾パターンの変化を、N末端側の23残基の部分に着目して横断的に解析する試みをしています。
論文に記載されていた、variable modificationパターンは以下の通りです。
- Acetyl (K),Acetyl (Protein N-term),Phospho (ST),Dimethyl (K), Methyl (K),Methyl (R), Trimethyl (K)
解析において特に着目した部分のペプチド配列は以下の通りです。
- SGRGKGGKGLGKGGAKRHRKVLR
修飾が付く可能性があるアミノ酸は S(1箇所1種類),K(5ヶ所4種類),R(4か所1種類) でさらにN末端にも修飾が付く可能性があります。今回のような多数の修飾組み合わせを考慮する解析において、MASCOT ver.2.7の初期設定値では不足があるのは明らかであるため、設定値の変更を行いました。
queryデータはMASCOT Distiller によってピーク抽出処理を行いました。MS/MSフラグメントイオンで多価のフラグメントピークについては1価相当のm/zに変換しました。得られたピークリストデータをMASCOT ver.2.6 並びにver.2.7 で検索し、その時に同定された修飾パターン (proteoforms と表記しています)数 をtable 3 にまとめました。
| Mascot Version | NumModifiedSites | NumVarMods | ModArrangements | No. proteoforms |
|---|---|---|---|---|
| 2.6 | N/A | N/A | N/A | 81 |
| 2.7 | 11 | 7 | 64 | 106 |
| 2.7 | 11 | 7 | 128 | 109 |
| 2.7 | 11 | 7 | 256 | 109 |
| 2.7 | 11 | 7 | 512 | 109 |
一目で差がわかるのが、ver.2.6に比べ ver.2.7 の時の方が、proteoforms数が増えている事です。加えて、ModArrangementsの値は 64より128にしたほうが良いことも確認できます。
さらに、ver.2.6ではうまくいかなかった修飾の同定がver.2.7ではうまくいった例についてもご紹介します。
Fig.1は、MASCOT ver.2.6 で一見なかなか良いマッチング(スコア112)をしているデータです。17番目と19番目のアルギニンがメチル化されているという事でレポートされていますが、マッチングを見ると該当箇所について理論値とスペクトルデータのマッチがありません。実際、生体内でもこのような修飾はありません。MASCOT ver.2.6 で採用している sliding windows のアルゴリズムが良くない結果となってしまった可能性の高い例です。
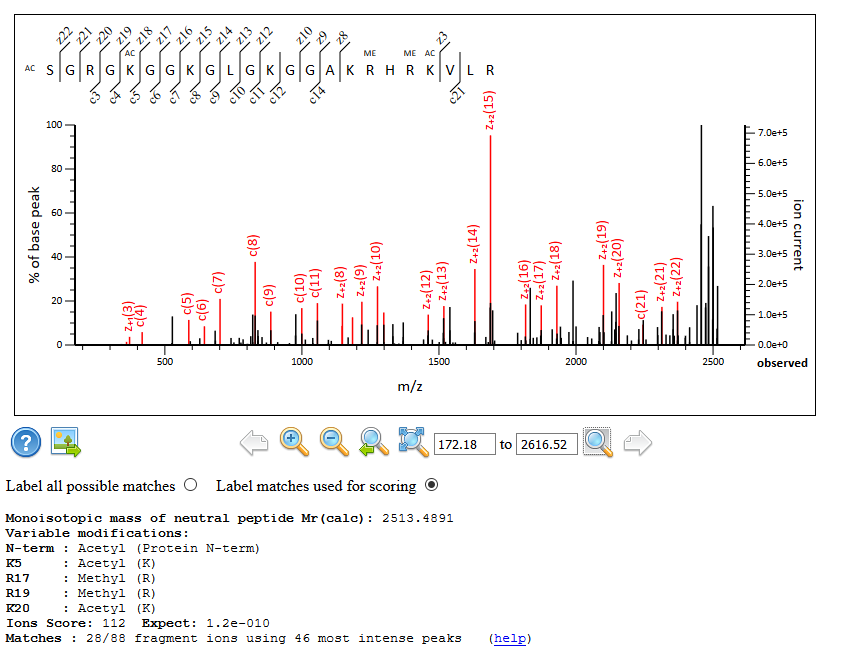
full size リンク
Fig.1 ver.2.6での解析例
一方 Fig.2 は 同じデータのMASCOT ver.2.7 の解析結果です。16番目のリジンがアセチル化、20番目のリジンがジメチル化、とリポートされています。先ほどの例と異なり該当箇所のピークも検出され、このような修飾は実際に生体でも起きることが報告されています。
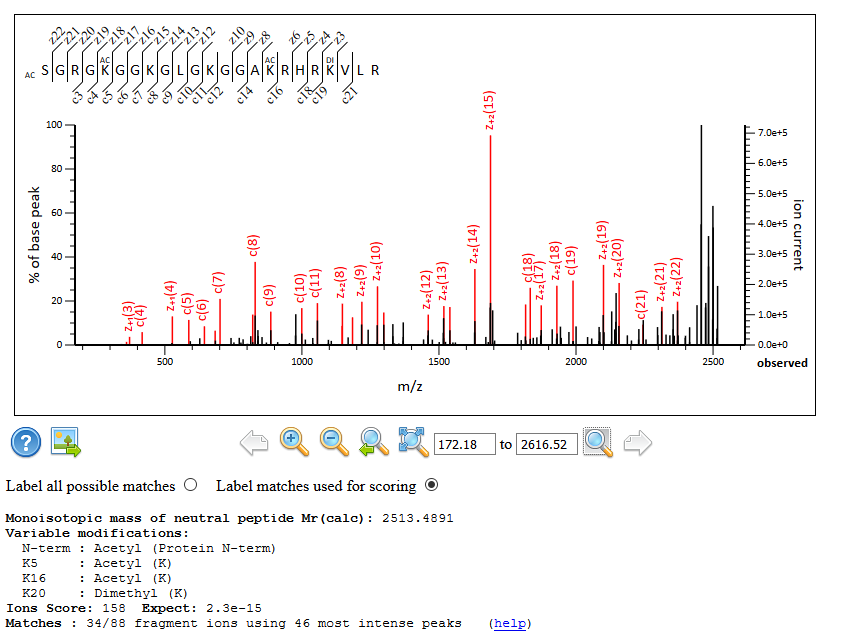
full size リンク
Fig.2 ver.2.7 での解析例(Fig1のqueryデータと同じ)
このように、variable modification に関する設定値を変更し複雑な修飾パターンに対応させることもできます。
variable modificationの詳細については、HELP ページをご覧ください。また本ブログ記事と同様の発表を過去に行いましたが、そのときのプレゼンテーション資料も併せてご参照ください。
訳者 補足
修飾部位の特定は、ペプチド同定より一歩複雑な難しい問題です。MASCOT における修飾部位の特定は、以下タグsite anaysisをご覧ください。
また弊社取り扱い製品として、Scaffold PTMもございます。ご興味がありましたら弊社日本法人までお問い合わせください。
Keywords: error tolerant, modification, permutation, site analysis, variable modifications