Unipept と Mascot
環境プロテオミクスあるいはメタプロテオミクスでは、ターゲットとなる生物種が数百から数千混ざりあった状態であり、そういった状況においてタンパク質レベルのデータから何かしらの結論を導き出す事はとても複雑で大変な作業です。今回はこのような研究において有用で補完的なリソースになりうるUnipeptデータベースについてご紹介します。Mascotのタンパク質推定(protein inference)と組み合わせて使用することができます。
ヒトの腸のサンプルの解析例
以前のブログ「Identify proteins by more than ‘gut’ feeling」において、MASCOTを用いた人間の腸のサンプルの解析について議論しました。その時はヒト腸内細菌のドラフトゲノムと次世代シークエンサーデータから構築されたメタゲノムデータを対象のデータベースとして検索をしました。メタゲノムデータはドラフトゲノムがカバーできない検索空間のギャップを埋めることを目的として使用しました。
Protein Family Summary レポートでは、シェアペプチドの存在をもとにタンパク質をグループ化しています。シェアペプチドの多寡は、配列の相同性とある程度関連性があります。そのため以前の解析の結果でも、ヒトのタンパク質はやはり細菌のタンパク質とは別のファミリーに分類されています。またバクテリアにおいては、ファミリー19のrubredoxinとrubrerythrinのように、互いに役割が類似しているタンパク質は生物種の枠を超えて同じファミリーにクラスタリングされています(上のスクリーンショット)。メタゲノムでのみ発見されたペプチドは、ドラフトゲノムのタンパク質とクラスター化することもありますし、「one hit wonder」、すなわち1つのペプチドのみが同定されていてタンパク質の同定としては少し確度が下がってしまうような状況、でリストに表示されているケースがあります。
Protein Family Summaryの結果では、生物の分類学的な概要がわかりにくいです。それを克服する助けとなるのが、Unipeptです。
Unipept
Unipeptはブラウザインターフェースを持つWebアプリケーションです。このデータベースは、UniProtKB内のすべての理論的なトリプシン断片を、Lowest Common Ancestor,(最低共通祖先,LCA)と紐づけしています。ペプチドのLCAは、Hymenobacter のVVSTNDANYRのケースのように単一種である可能性もありますし、逆にAELENTDSDYDRのように、異なる系統をまたがり多くの種の間で共有されていて、'Bacteria'などのようにグループ化され表現されている例もあります。さらに最近Unipept Desktopというアプリケーションがリリースされ、これまでよりも気軽にUnipeptを使用できるようになりました。特に入力データが大きくなってしまうようなケースで威力を発揮します。
今回MASCOTとUnipeptを組み合わせて使用する1つの例として、Mascot検索からすべての同定ペプチド配列をインポートし計算する方法についてご紹介します。Protein Family Summaryで、FDRを1% (スペクトル単位のカウント)に設定し、結果をCSVとしてエクスポートします。出力時に、エクスポート時のオプションである「same-set protein hits」のチェックを外し、「Include sub-set protein hits」の値を0に設定し、「Max number of hits」がAUTOに設定されていることを確認してください。出力されたCSVファイルには、すべての同定ペプチドが含まれています。これをExcelで開き、まずはpep_rank列でソートし値が1を超える行(rankが1位でないデータ)を削除します。その後、pep_seq列でソートして、ペプチド配列をテキストファイルにコピーします。このテキストファイルをUnipept Desktopで入力データとしてimportして利用してください。(その際、重複がフィルタリングされていることを確認してください)。今回のデータの大きさなら数分で解析が完了すると思います。
今回準備した入力データでは、2989のユニークな同定ペプチド配列が含まれており、そのうち2860はUnipeptにも含まれていました。Unipept Desktopは、MASCOT側で行っているタンパク質推定に該当する内容をLCAマッピングに置き換えてくれます。アプリケーションの計算により以下スクリーンショットのツリービューのようなまとめ図が作成されます。線の太さは各分類レベルでのユニークなペプチドのエビデンスの量を示しています。Chordata(脊索動物)のペプチドはデータを掘り下げてみるとウシのトリプシンやヒトのタンパク質につながっており、バクテリアはヒトの腸内細菌のタンパク質につながっています。ソフトウェアには様々な階層的なアウトライン、ヒートマップ、Gene Ontology情報を表示するオプションが用意されています。
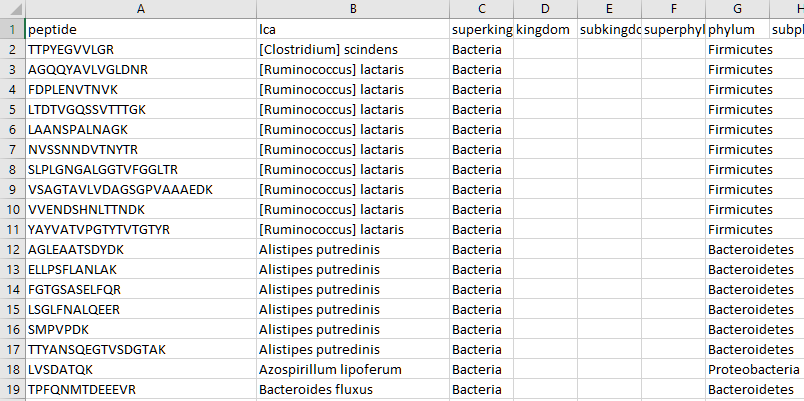
Unipeptの結果をCSVとしてエクスポートするだけでも多くの知見が得られます。出力データを’species'カラムでソートすると、以下のスクリーンショットに示すように、少なくとも1つユニークな同定ペプチドが存在するすべての種を一覧表示させることができます。
ところで129個のUnipeptにアサインされなかったペプチド配列ですが、それらはドラフトゲノム/メタゲノム にのみユニークに存在するペプチドということになります。今後より多くのゲノムが配列決定されUniProtKBに追加されれば、基本的にはUnipeptにも登録されていく事になるでしょう。例外のケースとして、非トリプシン断片にマッチしたペプチドというのも存在します。HTTQDHTTTPVLDHVNAAATANは翻訳されたORFにおけるC末端ペプチドであり、スコアも高いです。Mascotはまた、Bacteroides uniformisのhypothetical protein(訳者注:存在が確定的でない、ORFから仮定したタンパク質、の意味)から、Unipeptで規定しているよりも長いトリプシン断片ペプチドである、HTTQDHTTTPVLDHVNAAATANSISTDYNEAYFHVSPSVGVRとの一致を見つけています。もしサンプルに多くのトリプシン断片でないペプチド、または半特異的なペプチドが含まれている場合、(Unipeptでカバーできていない可能性を)心に留めておく必要があります。
ファミリーレベルの分析
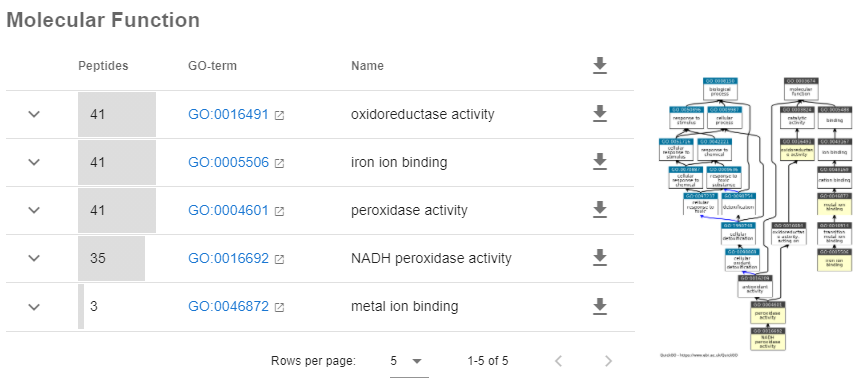
Unipeptはペプチドを直接生物種分類の情報やGene Ontology 情報にマッピングしているのですが複雑な構成になりがちで、例えば、そこからすべてのrubredoxinに関係するタンパク質のリストを取得するのは大変です。Unipeptで個々のペプチド配列を見ることで同定タンパク質の情報を見るために掘り下げていくことができますが、それよりは最初からMASCOTの結果のタンパク質ファミリーを限定し、そのペプチドだけを再解析する方がよほど簡単です。例えばMASCOTの結果を出力したCSVファイルからprot_hitが19(訳者注:rubredoxinのファミリー)であるペプチド配列を選択し、テキストファイルに保存し、これをインプットデータとしてUnipeptで新しいアッセイを作成することで、rubredoxinに関係するデータのみを集めた解析結果を見る事ができます。以下のスクリーンショットは、プロテインファミリー19(rubredoxin)に関連するGO terms の項目をまとめた図です。
Unipeptにはスクリプト可能なAPIがあります。これまで説明してきた、MascotのCSVエクスポートされたファイルを加工するなど、自動化することは難しくないでしょう。
現在のところ、Unipeptには解析に使用する種の範囲を制限する(フィルターをかける)仕組みはありません。例えば、データベースで生物種の対象が予め限定されていたMascotの検索結果においては、EGYPEVGLYYEKはDorea longicatenaのタンパク質rubredoxinにユニークにアサインされているペプチドです。しかし、この配列をUnipeptのトリプシン断片解析に貼り付けると、135個のタンパク質のリストが表示されます。これらが属する生物種の多くは通常人間の腸内では見られない種です。将来的には生物種を限定してデータベースの検索ができる機能が付与されるかもしれません。
Keywords: export, metaproteomics, protein inference