NCBIprot(NCBInr)の利用がMASCOT Server 2.8.1 で改善されました
日本では2022年4月4日にMascot Server 2.8.1パッチプログラムがリリースされました。大きな改善点として、NCBIprot(NCBInr)データベースの圧縮速度向上と最新バージョンが利用可能になった事が挙げられます。データベースをダウンロードしてから利用可能にするまでの時間が大幅に短縮されました。同時にNCBIprot構築に問題を引き起こしていたデータベースサイズに関する制限も削除しました。
NCBIprot(NCBInr)のサイズ
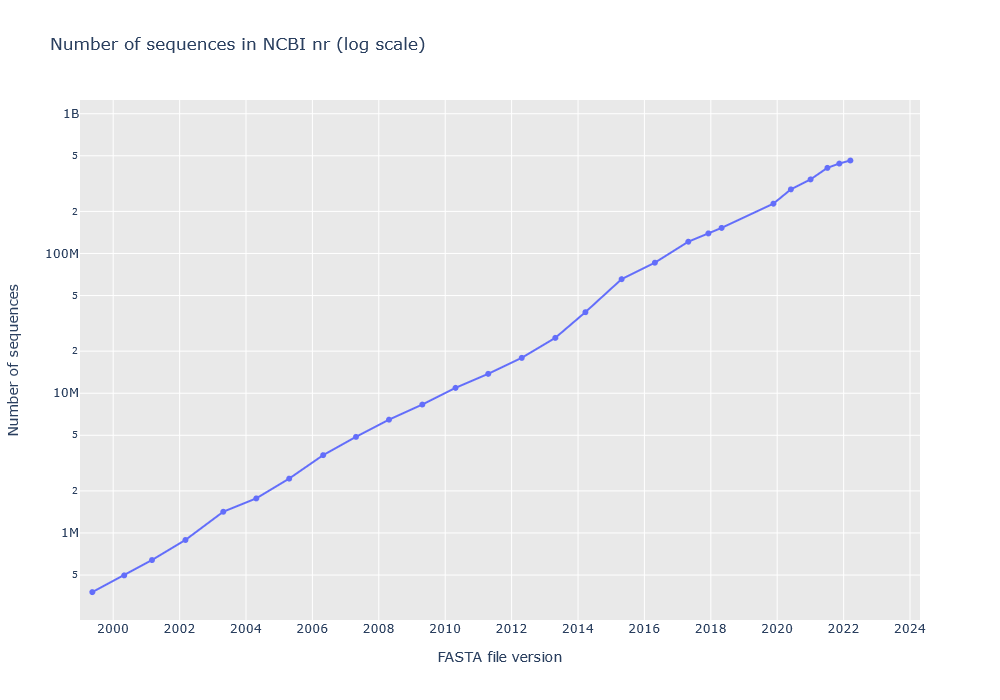
NCBIprot(NCBInr)はおよそ2年ごとにエントリー数が倍増しています。このデータベースはRefSeqやGenBankといったNCBIで公開されている複数データベースが合わさったタンパク質配列データベースで、新たに登録されるコンテンツは主にRefSeq由来のようです。
nrのタンパク質配列数の推移を表す公式なグラフはありません。 下記の時系列グラフは、弊社公開サイトの検索ログに含まれる情報から作成しました。 Mascotの結果ファイルのヘッダー領域にはFASTAファイルのバージョンと配列数が保存されています。弊社試用版公開サイトでは 1999年以降、NCBIprot(NCBInr)を検索可能なデータベースとして提供しています。
グラフは片対数目盛で、傾きはこの20年間同じです。このペースで行けば、2024年か2025年には10億配列に到達するはずです。これだけ大きなデータベースですから、ハード的にもソフト的にもあらゆる限界にぶつかる事でしょう。考えられる可能性とそれに対する弊社の取り組みを以下にご紹介します。
データベースサイズの制限 : 解決済み
MascotがFASTAデータベースを検索可能な状態にする際、いくつかの圧縮インデックスファイルを作成します。その目的は、タンパク質配列の高速ストリーミングを可能にすることです。インデックスファイルの1つ、拡張子が「.t00」のファイル は、各エントリーのTaxonomy(生物種情報)を格納しています。NCBIprot(NCBInr)は タンパク質配列が複数のアクセッションを持つ可能性がある構成になっている数少ないデータベースの一つです。そのため各タンパク質エントリーが複数のTaxonomy(生物種)情報を持つことがあります。MASCOTでは、各タンパク質のファイル位置(別名オフセット)をインデックスファイルに格納するデータ構造を採用しています。次のデータベースエントリーに移動する際、オフセット値に基づいてファイルハンドルが素早く転送されます。
これまで.t00のオフセットには32ビットの整数型を使用していました。そのためインデックスファイルのサイズが4GBを超えるとファイル位置が折り返されてしまい、挙動がおかしくなってしまいます。よくあきた問題としてデータベース検索が終了せず最終的にクラッシュしてしまう問題が報告されていました。
問題発生に伴い弊社で行ったNCBIprotでのテストによると、Mascot 2.8.0以前は3.39億の配列エントリーを扱えましたが、4.1億の配列エントリーは扱えませんでした。この2つの数字の間に境界線がある事を見つけるのにあまり時間はかかりませんでした。大きな問題であるこのバグについて、根本的な原因を突き止め不具合を修正する事の優先度は高かったからです。より正確には、このバグは配列数ではなく、関連するアクセッションの数によって引き起こされます。 nrは現在、1エントリーあたり平均約1.8アクセッションが紐づけされています。以前のバージョンのMascotでは最大で生物種セッション数が約6億5000万のFASTAファイルを扱うことができました。なお、生物種との紐づけを定義していないデータベースについては、.t00インデックスファイルを使用しないため、ファイルのサイズが非常に大きくとも今回の問題は発生しません。
Mascot 2.8.1では.t00ファイルのオフセットが64ビット整数型になり、.t00ファイルの最大サイズは8エクサバイト(8192ペタバイト)となりました。 NCBIのnrが2年毎に倍増すると仮定すると、2044年には 1兆の配列エントリーに達します。.t00ファイルはだいたい16TBくらいとなるでしょうが、それでも新しい仕組みにおける制限を大きく下回り問題なく使用できるはずです。1兆の配列エントリーのFASTAファイルはその大きさが470TB程度になる可能性があり普通には扱えないサイズとなりますので、NCBI側がそれまでにファイルを界や門レベルなどで分割するでしょうし、そういった意味でも新しい.t00サイズが制限に達することはないでしょう。
圧縮速度 : 解決済み
もう一つの問題は、インデックスファイルを作成しNCBIprotをMASCOT用に変換(圧縮)するためにかかる時間です。nrがもっと小さかったずっと昔、圧縮にかかる時間はデータベースサイズと比例関係でした。しかし、近年この関係は崩れ、より時間がかかるようになっています。
NCBIprot(NCBInr)データベース構築には、prot.accession2taxidを補足情報として利用しています。これはタンパク質のアクセッションをTaxonomyのIDに紐づけするために必要な情報が含まれるファイルです。さらにMASCOTでは情報を整理しよりコンパクトな内容にまとめたファイルprot.av2taxidを準備して使用しています。これはprot.accession2taxidを基に弊社が独自に作成しているファイルで、Taxonomyとアクセッションとの紐づけにおける特殊性に対応するために使用されます。データベース圧縮の際ほとんどのインデックスファイル はFASTAの並び順そのままの順番に対応した内容で作成され、メモリーもほとんど必要としません。しかし生物種情報の探索は別です。FASTA ファイルの複数アクセションに対応する生物種情報は、prot.accession2taxidやprot.av2taxidの並びに関係なく存在し逐一全範囲を対象に探索しなければなりません。それに対応するべくprot.av2taxid テーブルはランダムアクセス探索的に利用可能である必要があります。
Mascot 2.8.0以前のバージョンでは読み取り専用のデータベース.cdbを作成し高速なランダムアクセス検索を可能としていました。テーブルの内容は複数個に分割しメモリマッピングしています。分割cdbによる対応は、prot.av2taxidが非常に大きくなってしまい分割数が非常に増えてしまうつい最近まで特に問題ありませんでした。コンピュータサイエンスの用語を交えていえば、ランダムアクセスの探索時間はO(1)(一定時間、の意味)であるべきです。しかしこの数年エントリー数が非常に増えるにつれ、徐々にO(n)(線形時間)となり、ディスクアクセスのボトルネックの影響度が強まりました。
Mascot 2.8.1では新たな方針を採用し、prot.av2taxidをアクセッション文字列の最初の文字で水平分割する要素を追加しました。これによりランダムアクセスの探索時間は再びO(1)となるはずで、実際にベンチマーク結果でも確認されています。.cdbファイルは引き続きメモリー上にマップされます。利用可能なRAMが多ければ多いほど、OSがメモリにキャッシュしておける探索用のファイルが増え、高速になります。
推奨ハードウェア
前回のNCBIprot(NCBInr) tips の記事(英語版、日本語版)では、ディスクの性能の影響について簡単に説明しましたが、具体的な数値は示しませんでした。今回はいくつかのハードウェア構成における、構築にかかる時間について以下の表にまとめています。
| Type | CPU | RAM | Disk | Platform | NCBIprot compression 2.8.0 and earlier, 339M sequences |
NCBIprot compression 2.8.01, 440M sequences |
|---|---|---|---|---|---|---|
| Server | Intel Xeon 3.4GHz | 128GB | RAID10, 10k rpm HDDs | Linux | 5h | 6h |
| Workstation | Intel Core i7 3.2GHz | 64GB | SSD and HDD | Windows | 240h | 21h |
| Virtual machine | Intel Xeon 2.5GHz | 32GB | Virtualised disk on RAID50 host, 10k rpm HDDs | Linux | cancelled, predicted to take >1 week | 23-25h |
| Laptop | Intel Core i7 2.4GHz | 16GB | SSD and HDD | Windows | cancelled after 1 week, infeasible | 52h |
2.8.1ベンチマークでは2021年11月のnr FASTAを使用しており、このFASTAには 4.4億の配列が含まれています。一方2.8.0ベンチマークは2021年1月のFASTAを使用し、配列数は3億3900万と数が違います。新バージョン(2.8.1)ではすべてのケースでデータベース構築が成功しました。 一方旧バージョン(2.8.0)では、ワークステーションでも実用レベルと言えないほど非常に遅く、それ以下のスペックである仮想マシンやラップトップでは待てど暮らせど構築完了の兆しを見出すことはできませんでした。
構築速度に関連して最も重要な要素は2つ、RAMの量とディスクのアクセス速度です。10,000 rpmのディスクを使用したRAID10アレイでは、圧縮時間が最も短くなります。圧縮時間は他のシステムと比べ最短で、(訳者注:エントリー数の違いを考えると) 新旧のバージョンに大きな差はありません。利用可能なRAMの量を減らすと圧縮速度が低下し、ディスク速度がボトルネックとなり遅くなります。 なおRAIDとの差に比べれば、HDDとSSDの間にある構築時間の差はほとんどありませんでした。目安のメモリサイズとして、prot.av2taxid ファイルの 2 倍を大きく上回る容量を用意する事をお勧めします。現状では32GBでも十分という事になりますが、近い将来のことを考えれば64GBの方が明らかに良いでしょう。ただし今回のアップデートにより、たとえメモリーが少なくともNCBIprotを構築する事ができるようになりました。ラップトップコンピューターの例のように、(現状サイズなら)2-3日以内に構築可能です。
MASCOT ver.2.7 以前でNCBIprotを使用する場合
Mascot 2.7以前のバージョンでNCBIprotを使用したい場合、以下の2つの方法がご提案可能です。
最初の選択肢は、最新のFASTAファイルをダウンロードしそれを少なくとも2つに分割する方法です(訳者注: 巨大なファイルを分割する具体的な操作方法についてはご自身でご検討ください)。それぞれを別々のデータベースとしてセットアップします。構築方法はシーケンスデータベースセットアップヘルプをご参照ください。検索時に分割した両方のデータベースを選択して検索すればOKです。nrの最新バージョンを利用したい場合はこの方法で実施してください。ただし巨大なデータベースに対して検索をするため、より高性能なハードウェアが必要になります。最低でも128GBのメモリー、ディスクのシステムとしてはRAID5またはRAID10の構成が必要です。
もう1つの選択肢として、以前のバージョンのFASTAファイルを設定する方法をご提案します。 2021年7月以前のものはMascot 2.8.0以前のバージョンでも動作するはずです。 メリットは、あらかじめ定義された設定と1つのFASTAファイルを使用できることですが、この古いバージョンに今後も縛られることになります。なお、もし、適切なFASTAファイルをお持ちでない場合は弊社までご連絡ください。
訳者補足:
今回ご紹介したMASCOT ver.2.8.1 へのアップデートをご希望される方はこちらをご覧ください。なおアップデート後にすべての登録データベースを再構築するため準備完了までお時間がかかります。
保守にご加入のお客様はお伺いして弊社技術担当によるアップデート作業を実施させて頂く事も可能です。お気軽にお申し付けください。
Keywords: database manager, Fasta, pc hardware