Mascot Server 3.0における、予測RTとフラグメント強度情報
Mascot Server次期バージョンのリリース候補が試用版WEBサイトにて公開されています。新バージョンのリリースノート(仮)にもありますように、搭載予定の機能の一つとして、MS2Rescoreとの統合を含む、機械学習による結果の精度向上があります。今月の記事では、その機能について少し掘り下げてご紹介します。私たちがベータテストを実施している間、この記事をご覧になってお待ちください。
MS2Rescoreとは?
現在Mascot ServerにはPercolatorが搭載されています。以前のブログ記事「機械学習による再スコアリングはどのように機能するのか?」(日本語版、英語版)でも、Percolatorを使用した再スコアリングの原理について説明しています。 Mascot Server 3.0ではソフトウェアをより良いものとするため、予測機械学習、特に保持時間とフラグメント強度の予測、の機能を取り入れました。弊社独自にそれらの開発をするのではなく、すでに検証されて広く受け入れられているものを組み込むことを選択しました。
これらのアルゴリズムを組み込んだ解析をする上での問題は、多くの再スコアリング・パイプライン(一連の解析の流れ)から選択をしなければならない事でした。MCP 23(7), 100798 (2024)では最新のパイプライン12個を比較しています。どのパイプラインも独自の機械学習モデルを持っており、そのほとんどは互換性がありません。またほとんどのパイプラインはオープンソースであり、理論的には誰でもパイプラインをダウンロードしてインストールし、データベースの検索結果をパイプラインに渡して再スコアリングすることができます。しかし実際に本当に利用しようとすると、気の遠くなるほど難しい事もあるでしょう。適切なパッケージを探し出し、適切な互換性のある内容を選んでインストールしたり、その前にシステムのセットアップ(WindowsでDockerを有効にするなど)が必要であったり。検索エンジンの検索結果から、適切なファイルフォーマットによる出力ファイルを取得し、パイプラインを設定する必要もありますし(パラメーターも理解しなくては!)、DLLがなければ補い、時にはソフトウェアが稼働時にクラッシュしたら対応し、パイプラインの出力を、自身が利用する際に適切な形式に加工する必要があります。これらはすべて、本来なら研究とは関係ない、避けたいハードルです。
私たちの目的はシンプルです。簡単な操作で、MASCOTユーザーがよりよい検索結果を得る事です。今回採用したMS2Rescoreはゲント大学で開発された、AIがアシストするペプチド同定の再スコアリングモジュール並びにプラットフォーム("Modular and user-friendly platform for AI-assisted rescoring of peptide identifications ")です。 MS2Rescore はMascotの一部としてインストールされ、処理を繋ぐためのすべての設定は自動的に行われ、Mascot側が自動的にパイプライン用のデータのフォーマットと結果の読み出しを行います。その計算を経た結果はProtein Family Summaryに表示され、Mascot CSV、mzIdentML、mzTabなどの標準フォーマットを使用してエクスポートする事もできます。
MS2Rescoreは数年前から存在し、免疫ペプチドのような「解析が比較的困難な」データセットにおいて、感度(訳者注:この場合、同定基準を満たす同定ペプチド数)を大きく向上させることが示されています(Declerc et al. MCP 21(8), 2022)。 その効果は特に免疫ペプチドに限定されるものではなく、どのようなデータセットにも使用する事ができます。
MS2Rescore には2つの予測システムが組み込まれています。DeepLCは ディープラーニングを用いた(修飾を受けたケースも含む)ペプチドの保持時間の予測、MS2PIP は複数のフラグメンテーション方法、測定装置、ラベリング技術に対応した、高速で正確なペプチドフラグメントスペクトルの予測を行います。MS2PIPは、PrositのようなCEに依存するツールとは異なり、様々なコリジョンエネルギーで動作することが示されています(Gabriels et al、Nucleic Acids Research 47(W1), 2019)。
Yeast example, CPTAC study 6
Mascot Serverには、小規模な酵母のサンプル:CPTAC study 6の検索結果が含まれています。サンプルには、酵母のバックグラウンドにスパイクされたSigma UPS1タンパク質が含まれています。 この検索は小規模(8675スペクトル)ですが、データが高品質であるためサンプルとしては大変良いものです。世界中で行われているような単純なルーチン分析ですが、以下に示すように適切な機能拡張をソフトウェアに適用する事により、その結果が改善します。
このファイルは、Thermo LTQ-XL-Orbitrapで4時間のグラジエントを用いて取得されました(完全なSOPについてはPaulovich et al., MCP, 9(2):242-254, 2010の補足情報をご覧ください)。rawファイルからMascot Distillerを使ってピーク抽出しました。 50個のSigma UPSタンパク質をデータベース(Sigma_UPS)として設定し、コンタミナントデータベース、Sigma_UPS、S. cerevisiaeプロテオームデータベースの3つを併せて検索対象としました。検索結果はこちらで確認する事ができます。
以下の表は、Mascot Server 2.7、2.8、3.0を各バージョンで利用可能な最良の設定を適用し検索した結果を比較したものです。検索のパラメーターとMGFピークリストの中身はすべて同一です。 修飾は固定がカルバミドメチル化(C)、可変が酸化(M)と設定した、非常に単純な検索です。
| Mascot Server | Unique sequences | Sequence FDR | Protein hits | Protein FDR | Capabilities | Limitations |
|---|---|---|---|---|---|---|
| 2.7 | 2702 | 0.59% | 759 | 1.84% | Percolator 3.0; unoptimised features | Cannot get to 1% sequence FDR |
| 2.8 | 2768 | 0.98% | 763 | 3.15% | Percolator 3.5; optimised core features | Cannot use predicted features |
| 3.0 (beta) | 3431 | 0.99% | 863 | 3.36% | Percolator 3.5; optimised core features; MS2Rescore, DeepLC, MS2PIP |
上記の数値は、同定基準として1% sequence FDRを適用しており、1% PSM FDRよりも厳しい閾値です。Mascot Server 3.0 よりsequence FDRがデフォルト設定となります。
Mascot Server 2.7でPercolatorを有効にした場合、フォーマット制御におけるsigthresholdパラメーターに様々なバグがあったため、1%の sequence FDRぴったりに調整する事ができませんでした。私たちはMascot Server 2.8 にアップデートする際、Mascotが計算するコアフィーチャーの最適化に多くの時間を費やしました。 その結果、内因性ペプチドのような難しいデータセットでは改善が見られました。一方今回のように単純な酵母検索においてはあまり結果が良くなりませんでした。
今回 Mascot Server 3.0で採用した予測機械学習は、とても素晴らしい効果がありました。検索において選択したモデルは以下の通りです:
DeepLC: 「full_hc_unmod_fixed_mods」。トレーニングデータが4hグラジエントのトリプシン性酵母ペプチドであったためです(DeepLCモデルの全リスト)。 CPTACデータは135分のグラジエントで取得されており、4hグラジエントのモデルはよくマッチしています(さらに記事の下部も参照してください!)。
MS2PIP:「CID」。CPTAC酵母のデータがLTQ-XL-Orbitrapで取得されたためです(MS2PIPモデルの全リスト)。「CID」モデルはNIST CID Humanスペクトルライブラリを使ってトレーニングされています。このスペクトルライブラリは、様々な装置からのCIDスペクトルを、非常に高品質で精錬したコレクションです。
正しく動作している事をどうやって確認するか?
機械学習は魔法ではありません。うまくいかない事もあります。この最先端の技術は与えられたペプチド配列と可変修飾に対して常に予測を与えますが、間違った予測をすることもあります。
Mascot Server 3.0には、機械学習の品質レポート機能があります:正しいモデルを選択したかを判断する一助となります。リンクはProtein Family SummaryのSensitivity and FDRセクションにあります(酵母+UPSデータの例)。
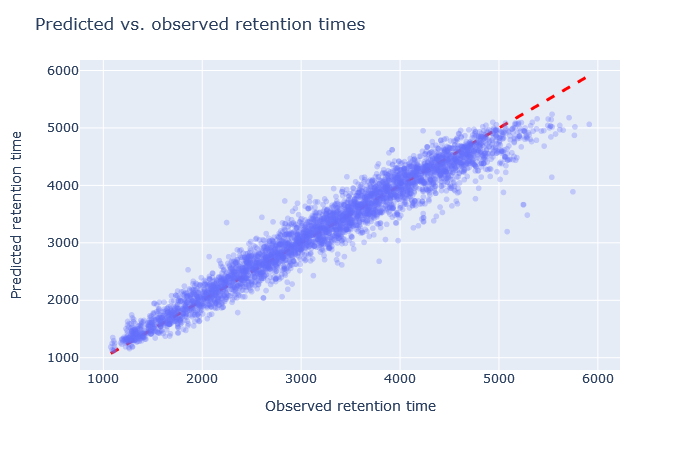
今回ご紹介した酵母データセットにおける、予測された保持時間と観測された保持時間の散布図は上図のようになっていて、予想が的確である事がわかります。散布図はターゲットデータベースの有意なマッチのみを示しています(おとりマッチではありません)。外れ値はどのようなデータでも常にありますが、重要なのは対角線に沿ってしっかりと束になってデータが存在するかどうかです。MS2PIP、CIDモデルが使用された今回のサンプルは、予測値が中央値で相関0.88となり非常に良好です。他にも多くの点について機械学習の品質レポートから確認する事ができます。その中身を見ると、このデータセットに対して選択したモデルが非常に適切であったと判断できます。
1%FDRで感度(訳者注:この場合は同定ペプチド数)が向上した理由は、予測保持時間との差が大きい不正確なマッチが半教師付き機械学習によって抑制されたからです。予測保持時間が観測保持時間に近い正しいマッチはブーストされます(訳者注:極端に言えば、保持時間予測が近い場合、スペクトルマッチが多少悪くても同定と判定する、と考えてよいかと思います)。
Masco Server 3.0は現在ベータテスト中です。リリースの準備が整い次第、ニューズレターでお知らせします。また利用権のある方にはDVDとライセンスを発行いたします。今しばらくお待ちください。
Keywords: machine learning, Percolator, scoring