Mascot vs FragPipe: 内因性のタンパク質分解の解明
内因性のタンパク質分解、またはN末端プロテオミクス(N-terminomics)研究では、通常、タンパク質のN末端を選択的に濃縮します。 しかし濃縮をしないもう1つの方法として、標準的なショットガンLC-MS/MSアプローチを適用する事ができます。この場合、切断箇所が半特異的なペプチドを同定するデータベース検索が必要です。Mascot Server 3.0 にはフラグメント強度予測のための機械学習モデルである MS2PIP が含まれており、これにより半特異的切断のペプチド同定が大幅に改善されます。FragPipe の最新バージョンも同様の再スコアリングの考え方を採用しており、MSBoost により予測を行います。この記事では、両検索エンジンで行った検索結果の比較を行います。
マウスの多発性腎嚢胞(PDK)モデルにおける末端プロテオミクス(terminomics)
比較のためのデータとして、MassIVE プロジェクト MSV000094661 からrawデータを取得しました。これは、内在性タンパク質分解プロセシングを分析するパイプラインであるTermineRの最近の論文で使用されたデータです。公開されているrawデータの中で、論文の2番目の実験データを選択しました。この実験では、末端プロテオミクスの(terminomic)マウス多発性腎嚢胞症(PDK)モデルのTMTpro標識試料を処理しています。腎嚢胞の肥大が見られた腎臓組織サンプルが6つ、野生型サンプルが5つのデータで構成されています。
TMTproラベル化はタンパク質レベルで行われたため、N末端ペプチドのラベルについて、可変な修飾としてパラメーター指定する必要があります。消化前にサンプルを標識することで、遊離タンパク質のN末端部位はすべてTMTproで標識されますが、その同定結果から、「予期せぬタンパク質分解」が明らかになります。
N末端ペプチドは必ずしもトリプシン切断の規則に従うとは限らないため、切断パターンが”半特異的”である事を考慮するパラメーターを使用する必要があります。また、トリプシンはTMTpro標識Kを切断することができません。そこで弊社では、半特異的な切断を行うArgC酵素を作成し検索に利用しました。また、想定外のタンパク質の切断とN末端のアセチル化について特定するため、タンパク質レベル(Protein N-term)ではなく、ペプチドレベル(N-term)でN末端がアセチル化される設定を可変項目として選択しました。
検索には3つのデータベースを使用しました。1. Uniprot UP589_M_musculus proteome、2. contaminants(トリプシン、ヒトケラチン、その他の一般的なコンタミネーションを含む)、3. Biognosys_iRT peptide。
上記以外のパラメーターは、発表論文と同じ内容にしています。最終的な検索パラメーターは以下の通りです。
Enzyme : SemiArgC Fixed modifications : Carbamidomethyl (C), TMTpro (K) Variable modifications : Acetyl (N-term), TMTpro (N-term) Mass values : Monoisotopic Protein mass : Unrestricted Peptide mass tolerance : ± 20 ppm Fragment mass tolerance : ± 20 ppm Max missed cleavages : 1 Instrument type : ESI-TRAP
データを処理している過程で、スペクトル内にTMTproのcomplementary reporter ionのクラスターが多数存在することに気づきました。これを適切に処理するため、スクリプトTMT_complementary_ions.plを使用してcomplementary reporter ionをすべて削除しました。これにより、ペプチドのスコアリングと同定が若干改善されました。別のオプションとして、complementary reporter ionを通常のレポーターイオンとして変換する方法もありますが、今回は実験で使用されたTMTproラベルから不可能であると判断しました。一部のチャンネルが重複してしまうためです。
結果
Mascot Server 3.0 を使用して検索を行い、機械学習による検索結果の改善機能を有効にし、MS2PIP のパラメーターとしてTMTを選択しました。TMTpro のラベルは、通常のパターンと比べてフラグメンテーションパターンを大幅に変更するため、TMT以外の選択肢は標識ペプチドに適していません。
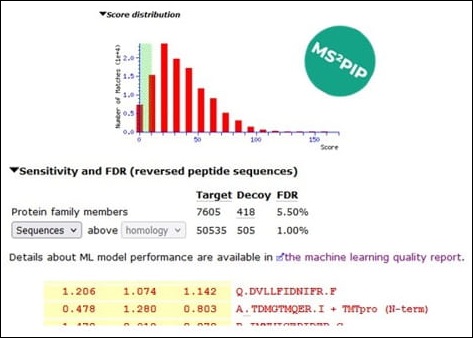
Mascotでは保持時間の予測のためのアルゴリズムDeepLCも搭載しており、その選択モデルの中には TMT向けのパラメーターも存在します(full_hc_hela_hf_psms_aligned)。しかし今回のデータについては十分な結果の改善が見られず、計算時間が延びただけでした。また、他のDeepLCの選択肢を選んでも、結果の改善は見られませんでした
| FragPipe with MSBoost |
Mascot with MS2PIP TMT model |
|
|---|---|---|
| Protein hits | 7560 | 7605 |
| Unique sequences | 44470 | 50535 |
| TMTpro N-term | 3062 | 3219 |
| Acetyl N-term | 617 | 883 |
FragPipe の結果の数字については、MassIVE リポジトリからダウンロードした .tsv レポートファイルから取得しました。スプレッドシートには 1% FDR に閾値調整された内容が記載されていると考えられます。
一方Mascot Serverの結果の数字については、レポートのFDR"statistics"セクションからを取得しました。結果はSequence FDR1%を基準としています。同定タンパク質は、ユニークなペプチドのアサインによる裏付けがあります。結果をCSVファイルに出力しました。
両方のスプレッドシートをExcelで開きさらなる計算処理を行いました。Excelでいくつかのフィルタリングとピボット(データのまとめ直し)を行う事で、N末端のアセチル化とN末端のTMTproのマッチング数をカウントしました。このうちタンパク質のN末端ペプチド(直前の残基が「-」と表記されている)については省きましたが、N末端残基がメチオニンであるペプチドであった場合は省きませんでした。メチオニンはタンパク質の1番目の位置し翻訳後の処理中に除去されるため、実際には該当ペプチドが実際のタンパク質の実質的なN末端となるはずです。対象として残したペプチドの大部分は、消化前の不完全なタンパク質のN末端から得られた半特異的ペプチドであると考えられます。
最後に、今回の解析においてMASCOT計算処理にはどのくらいの時間がかかるのかについてもご紹介します。rawファイルは約600,000件のクエリ(MS/MSスペクトル)に変換され、16コアをもつCPUのサーバー(AMD Ryzen 9 9950X)でMascot Serverによるデータベース検索を行ったところ、303秒かかりました(検索におけるパラメーターの切断パターンは"Semi-Specfic"です)。機械学習の計算にはさらに数分かかりました。半特異的酵素は検索空間を大幅に広げるものの、マウスのプロテオームにはわずか63,149のタンパク質配列しか登録されていないため、それほど莫大な計算時間がかかる事はありません。この登録件数は、Mascotが設計の際想定していたエントリー数と比べても非常に少ないと言えます。
Keywords: machine learning,MS2PIP, Percolator,TMT