MascotとProteome Discovererの連携において、機械学習機能を使用する
この度、Mascot Server 3.1をリリースいたしました。Thermo Proteome Discoverer™(以下、"PD"の略称を利用する事があります)との統合を強化すると共に、Mascot Server 3.0で発生したいくつかの重要なバグが修正されたマイナーアップデートです。 改善点・変更点の全リストはrelease announcement をご覧ください。 特に注目すべきはProteome Discovererとの統合利用において、Mascotにてrefinementされた結果を直接PDにインポートできるようになった事です。 この機能を利用する事で、従来に比べペプチド同定数が50%以上増加し、それに伴って同定されたタンパク質数も増加します。
Proteome DiscovererからMASCOTの機械学習アルゴリズムを利用するには
Mascot Server 3.0 では、2つの新しい検索パラメータが導入されました。 PERCOLATE と ML_ADAPTER_PARAM です。 これらのパラメータは通常、検索フォームまたは検索後の結果レポート上で設定します。 これらのパラメータを使用すると、結果の精査(refinement,PERCOLATE)が可能になり、さらにオプションとして、DeepLC または MS2PIP のモデル(ML_ADAPTER_PARAM)を選択することができます。
Mascot ver.3.0へのアップデート以降、Proteome Discovererでこれらのパラメーターを指定する方法がありませんでしたが、Mascot Server 3.1でその回避策が実装されました。それらの機能を”Instrument”パラメーターに組み込んで利用する事ができるようになりました。パラメーター名としては例えば「MS2PIP:HCD2021」などの名称で保存します。そしてPDから検索を実行する際には、Instrumentのパラメーターを適切に作成しそれを検索時にパラメーターとして選択するだけです。検索が終了すると、Mascotは機械学習により結果をrefinementし、その結果をPDに送信します。
この記事が書かれた時点では、PD 2.4、PD 3.1、PD 3.2での統合利用がテストされています。PDのAPIとデータ形式は長年にわたって安定しているので、おそらくPDのすべてのバージョン(1.4、2.x、3.0、3.1、3.2)で動作するであろうと考えています。
解析例:ヒト、酵母、大腸菌の混合サンプル(PXD028735)
PRIDEプロジェクト PXD028735から、論文 A comprehensive LFQ benchmark dataset on modern day acquisition strategies in proteomics (Pyuvelde et al., Scientific Data, 9(126), 2022)のrawデータが入手できます。著者らは6種類の装置を使用し、酵母、大腸菌、ヒト由来のタンパク質について、比率が異なる6種類の混合物を解析しました。あらゆるサンプル・あらゆる装置のDDAによる解析です。その中から私たちは今回Thermo Orbitrap QE HF-X(ナノフローLC)で測定されたQC繰り返し実験の1つLFQ_Orbitrap_DDA_QC_03.rawをダウンロードしました。このrawファイルを選んだ特別な理由はなく、任意に選択されたものです。他のrawファイルでも同様の結果が得られるでしょう。
Mascot Server 3.1で、新しいInstrument 設定である「MS2PIP:HCD2021」を設定しました。この設定の中で、refinementを有効にしMS2PIPモデルとしてHCD2021を選択しています。設定の作成手順については資料「Using machine learning with Mascot Server 3.1 and Proteome Discoverer」(4ページ、254 KB)を参照してください。
Proteome Discovererで利用するための手順についてもご説明します。まず、ConsensusWF \ CWF_Basic.pdConsensusWFテンプレートを使用して新規の”study”を作成します。processingワークフローでは、下図のようにMascotを設定します。
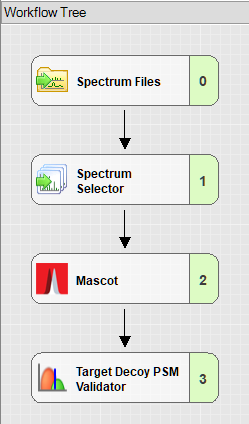
すなわち、Spectrum FilesをSpectrum Selectorに接続し、続いてMascotにデータを渡すように設定します。最後に、Target Decoy PSM Validatorで検証を行います。この時Target Decoy PSM Validator strategyのパラメーターをSeparateに設定してください。
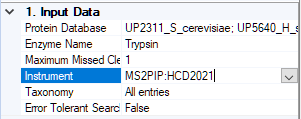
Mascot 検索では以下の検索パラメーターを使用しました。使用するデータベースのセットは、Mascot のDatabase Managerにおいて、”predefined definition”機能を使って素早く簡単に行う事ができます。
- Protein Database: UP2311_S_cerevisiae, UP5640_H_sapiens, UP625_E_coli_K12
- Enzyme Name: Trypsin
- Maximum Missed Cleavages: 1
- Instrument: MS2PIP:HCD2021
- Taxonomy: All entries
- Error Tolerant Search: False
- Precursor Mass Tolerance: 10 ppm
- Fragment Mass Tolerance: 20 ppm
- 1. Dynamic Modification: Oxidation (M)
- 1. Static Modification: Carbamidomethyl (C)
その他の処理設定などはデフォルト設定をそのまま利用しても構いません。
Instrumentの選択により、機械学習を使用するかどうかを選ぶことができます。例えば今回の例で使用する場合、Instrumentの設定でMS2PIP:HCD2021を選んでください。
PD 3.1では、ペプチドのマッチ数がほぼ60%増加
以下の表は、同定ペプチド数・同定タンパク質数について、HCD2021モデルとProteome Discoverer 3.1を使用した場合の効果をまとめたものです。
| Mascot | PD | Protein Groups | Peptide Groups | Threshold |
|---|---|---|---|---|
| 3.0 (no ML) | PD 3.1 | 4,635 | 21,507 | Expect value: 0.87 |
| 3.1 (with ML) | PD 3.1 | 5,898 | 34,141 | Expect value (PEP): 0.1256 |
有効な閾値はresult viewerから確認できます。PSM タブの画面でExpect Valueで並び替え、最大値を探してください。Mascot Server 3.0 でrefinement機能を無効にした場合と比較すると、1% FDR に調整した際の期待値は上昇します。すなわち、0.05 から 0.87 のあいだの疑わしい品質の多くのマッチングを受け入れなければなりません。
それに対してMascot Server 3.1 を使用しrefinementを有効にした場合は、より妥当な閾値が提示されます(0.1256)。Proteome Discoverer はより多くのマッチングを検出するだけでなく、そのマッチングは統計的により信頼性の高いものとなります。ただし一点ご注意いただきたいことがあります。両者で計算されている数値は厳密には同じ指標・数値ではありません。ver.3.1を使用した計算で [Expect] 列の値で示されている値は、実際にはPercolator によって計算されたposterior error probability(事後誤り確率 ,PEP)であることに注意してください。
PD 2.4 ではペプチドのマッチングが 100% 以上増加
Proteome Discoverer 2.4でも同じワークフローとrawファイルを使用して実行しました。
| Mascot | PD | Protein Groups | Peptide Groups | Threshold |
|---|---|---|---|---|
| 3.0 (no ML) | PD 2.4 | 4,235 | 17,106 | Expect value: 0.1675 |
| 3.1 (with ML) | PD 2.4 | 6,100 | 35,047 | Expect value (PEP): 0.09976 |
PD 2.4 と PD 3.1 の間に若干の違いが生じる理由は、PD タンパク質推論アルゴリズムと、各バージョン間の Target Decoy PSM Validator ノードの違いによるものです。この比較において、ペプチドグループの 同定数は100% 以上の大幅な増加となっています。これはMascot ノードが行っているスコアの閾値の違いが関係しています。
PD 2.4ではMascotノードにおいて同定基準値はidentity thresholdのみが使用されます。一方PD 3.1ではhomology thresholdが同定基準値に使用されます。Homology threhsoldは経験的に決定された閾値であり、通常はユーザーが指定したFDR(通常は1%)において、感度(この場合は同定ペプチド数)が大幅に向上します。今回の結果でも、PD 2.4が17,106であるのに対して、PD 3.1では21,507と、refinementなしの結果を比較しても明らかです。
Mascotが結果をrefinementした場合、「Mascotスコア」は10log10(PEP)として定義しています。またIdentity thresholdは20です。スコア20はPEP 0.01と同じ意味です。refinement実施後の結果についてPD 2.4(35,047ペプチドグループ)とPD 3.1(34,141ペプチドグループ)を比較すると、両者の違いはほとんどありません。
Keywords: machine learning, MS2PIP, Percolator, Proteome Discoverer